ProteinBLAST會(huì)成為過去嗎?AlphaFold等對(duì)蛋白結(jié)構(gòu)的搜索是否會(huì)取代對(duì)序列的搜索

編輯 | 白菜葉
像 AlphaFold 這樣的蛋白質(zhì)結(jié)構(gòu)搜索工具會(huì)用 BLAST 取代蛋白質(zhì)序列搜索嗎?德累斯頓工業(yè)大學(xué)的研究團(tuán)隊(duì)討論了使用結(jié)構(gòu)搜索進(jìn)行遠(yuǎn)程同源性檢測(cè)的前景,以及為什么蛋白質(zhì) BLAST 作為領(lǐng)先的序列搜索工具應(yīng)努力納入結(jié)構(gòu)信息。

BLAST 廣泛用于分子生物學(xué)中搜索核苷酸和蛋白質(zhì)序列。BLAST 推出三十年后,結(jié)構(gòu)預(yù)測(cè)出現(xiàn)了重大突破,出現(xiàn)了 RoseTTAFold 和 AlphaFold 等工具。
因此,主要序列數(shù)據(jù)庫(kù)中的每個(gè)蛋白質(zhì)序列現(xiàn)在都帶有一個(gè) 3D 折疊模型。雖然這不會(huì)影響(非編碼)核苷酸序列,但它引出了一個(gè)問題:對(duì) 3D 蛋白質(zhì)結(jié)構(gòu)的搜索是否會(huì)取代對(duì)蛋白質(zhì)序列的搜索。Protein BLAST 已經(jīng)成為過去了嗎?
雖然 BLAST 搜索是功能預(yù)測(cè)的強(qiáng)大工具,但它的能力是有限的。序列經(jīng)過處理可以顯著降解,但仍然會(huì)折疊成執(zhí)行相同或相似功能的類似 3D 結(jié)構(gòu)。
不同的序列,相同的結(jié)構(gòu)
這種蛋白質(zhì)對(duì)的例子可以在藻類和細(xì)菌的粘附分子中找到,特別是在硅藻粘附蛋白 CaTrailin_4 和細(xì)菌冰結(jié)合蛋白 FfIBP 中。該對(duì)沒有可通過 BLAST 檢測(cè)到的序列相似性(E 值 0.30,其中 E 值?>?0.001 不被認(rèn)為是顯著的)。
事實(shí)上,即使是更精細(xì)的基于序列的工具(例如 HHblits)也無法建立關(guān)系。然而,CaTrailin_4 的預(yù)測(cè)結(jié)構(gòu)和 FfIBP 的已知結(jié)構(gòu)非常相似,因?yàn)閮烧叨疾捎糜?α 螺旋持有的兩個(gè)單元組成的 β 螺旋折疊 - 冰結(jié)合蛋白的拓?fù)涮卣鳌?/p>
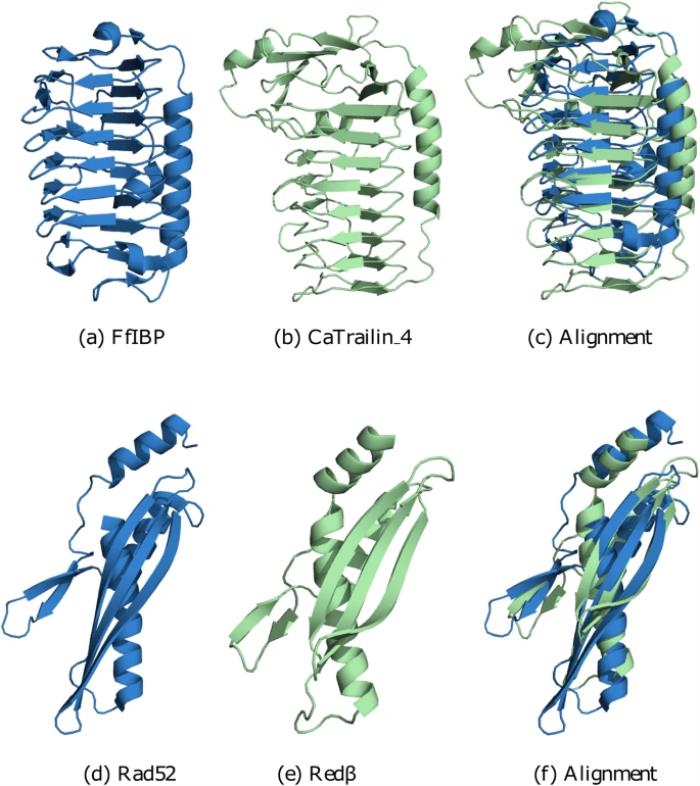
這種結(jié)構(gòu)相似性可以通過所謂的模板建模分?jǐn)?shù)(TM-score)來衡量,它結(jié)合了 RMSD(均方根偏差)和比對(duì)長(zhǎng)度作為可解釋的分?jǐn)?shù)。大于 0.5 的 TM 分?jǐn)?shù)意味著兩個(gè)結(jié)構(gòu)可能采用相同的折疊并且在進(jìn)化上相關(guān)。CaTrailin_4 和 FfIBP 的 TM 分?jǐn)?shù)為 0.6(高于 0.5 截止值)。因此,結(jié)構(gòu)比較可以揭示這種驚人的相似性,而這對(duì)于 BLAST 和其他基于序列的工具(例如 HHblits)來說仍然難以捉摸。
另一個(gè)例子涉及 DNA 重組,這是復(fù)制的基本過程,其中單鏈退火蛋白 (SSAP) 發(fā)揮著核心作用。二十多年來,RecT/Redβ、ERF 和 RAD52 是否形成三個(gè)不同的超家族,或者只是一個(gè)超家族,一直受到懷疑和爭(zhēng)議性的討論。前一種觀點(diǎn)得到了序列分析的支持,序列分析顯示 RecT/Redβ、ERF 和 RAD52 之間沒有明顯的相似性。事實(shí)上,Rad52 和 Redβ 沒有通過 BLAST 檢測(cè)到的相似性(E 值 0.38)。
考慮結(jié)構(gòu)會(huì)改變情況。Al-Fatlawi 團(tuán)隊(duì)將 RecT/Redβ、ERF 和 RAD52 的代表性結(jié)構(gòu)并列在一起,結(jié)果表明,盡管缺乏序列相似性,但這些結(jié)構(gòu)包含一個(gè)核心結(jié)構(gòu)元件。它是寡聚反應(yīng)的核心,因?yàn)樗謩e生成環(huán)和螺旋結(jié)構(gòu)。因此,它在 RecT/Redβ、ERF 和 RAD52 中非常保守,并且可以通過結(jié)構(gòu)相似性(TM 得分為 0.5)檢測(cè)到,盡管缺乏任何序列相似性(見圖 1 d-f)。
結(jié)構(gòu)預(yù)測(cè)來拯救
這些例子表明 AlphaFold 或許能夠介入 BLAST 無法發(fā)現(xiàn)顯著相似性的領(lǐng)域。因此,問題出現(xiàn)了:如何系統(tǒng)地實(shí)現(xiàn)這一目標(biāo)?為此,出現(xiàn)了 Foldseek、DALI 和 3D-AF-Surfer 等工具,它們分別使用自動(dòng)編碼器、距離矩陣對(duì)齊和專用指紋來掃描和比較結(jié)構(gòu)。
雖然這些工具已經(jīng)存在,但它們?nèi)匀恍枰訌V泛和簡(jiǎn)單,以便同序列數(shù)據(jù)庫(kù)上的 BLAST 搜索競(jìng)爭(zhēng)。需要協(xié)同作用將它們集成到經(jīng)典的 BLAST 序列搜索中。最近,一項(xiàng)研究比較了倒數(shù)最佳 BLAST 命中和倒數(shù)最佳結(jié)構(gòu)命中,并通過對(duì)序列的機(jī)器學(xué)習(xí)嵌入進(jìn)行最近鄰搜索,在這個(gè)方向上邁出了第一步。
為了探索這種先進(jìn)工具的潛力,研究人員想要了解同一超家族的成員資格標(biāo)準(zhǔn)如何與序列和結(jié)構(gòu)相似性聯(lián)系起來。因此,科學(xué)家曾從 SCOPe 數(shù)據(jù)庫(kù)中獲得了 11,211 個(gè)具有超家族的域。這些形成 62,278,380 個(gè)結(jié)構(gòu)域?qū)Γ渲?225,931 個(gè) (0.36%) 屬于同一超家族,因此可以被視為同源物。
這些同源對(duì)中有多少可以分別通過序列和結(jié)構(gòu)直接找到?在 E 值截止值為 0.001 時(shí),BLAST 從 225,931 對(duì)中恢復(fù)了 16,300 對(duì) (7%)。將界限放寬至 1,該數(shù)字增加至 25,634(11%)。但即使 E 值?< 10,也不會(huì)超過 15%。如果考慮更敏感的基于序列的方法(例如隱馬爾可夫模型),這些數(shù)字會(huì)大大改善。事實(shí)上,HHblits 在最佳條件下能夠檢索到 175,682 對(duì)(78%),這甚至比通過結(jié)構(gòu)比較(TM-score >?0.5)找到的 164,468 對(duì)(73%)要好。
然而,那 62,052,449 對(duì)不屬于同一超家族的呢?在這些對(duì)中,E 值小于 0.001、1 和 10 的對(duì)分別有 0 個(gè)、9,053 個(gè)和 72,329 個(gè)。HHblits 在這 25% 中進(jìn)行識(shí)別,而結(jié)構(gòu)對(duì)齊的錯(cuò)誤檢測(cè)被限制在 2% 以下。HHblits 的 AUC 為 77%,結(jié)構(gòu)比較為 95%,而 Blast 為 44%。較高的 AUC 分?jǐn)?shù)表明,與其他超家族中的蛋白質(zhì)相比,分類器能夠更有效地為正確超家族中的蛋白質(zhì)正確分配更高的分?jǐn)?shù)。
盡管結(jié)構(gòu)比較的 95% AUC 可能令人鼓舞,但高質(zhì)量結(jié)構(gòu)的可用性可能是一個(gè)限制。據(jù)估計(jì),30% 的真核蛋白質(zhì)含有 50 個(gè)或更多連續(xù)氨基酸的無序區(qū)域,這在 3D 結(jié)構(gòu)預(yù)測(cè)中預(yù)計(jì)質(zhì)量較差。這些區(qū)域適合使用 BLAST 進(jìn)行序列搜索,但不適合直接結(jié)構(gòu)搜索。
為了評(píng)估如此大的百分比如何擴(kuò)展到整個(gè) AlphaFold 數(shù)據(jù)庫(kù),研究人員計(jì)算了所有 AlphaFold 結(jié)構(gòu)的平均置信度得分。研究人員發(fā)現(xiàn) 80% 的 AlphaFold 結(jié)構(gòu)的 pLDDT 置信度得分為 70% 或更高,這意味著它們可以通過總體良好的主干預(yù)測(cè)進(jìn)行良好建模。這意味著存在大量質(zhì)量合適的結(jié)構(gòu)數(shù)據(jù)。
BLAST,未來之事
BLAST 完美地滿足了生物醫(yī)學(xué)研究人員的許多需求,例如檢測(cè)變異和密切相關(guān)的序列。然而,遠(yuǎn)程同源性檢測(cè)的具體問題對(duì)于純序列搜索來說是困難的。
在這里,結(jié)構(gòu)可以比順序更進(jìn)一步。研究人員通過對(duì)數(shù)百萬對(duì)結(jié)構(gòu)域的演示分析來評(píng)估序列和結(jié)構(gòu)相似性的這種關(guān)系。總而言之,分析表明具有嚴(yán)格 E 值的 BLAST 在尋找同源物方面非常精確,但并不全面。隱馬爾可夫模型更敏感,但特異性有限。結(jié)構(gòu)比較平衡了這兩個(gè)極端。如果 BLAST 搜索包含結(jié)構(gòu)數(shù)據(jù),它可以擴(kuò)展具有相似預(yù)測(cè)結(jié)構(gòu)并且可能是候選同源物的命中數(shù),而不會(huì)損害結(jié)果的質(zhì)量。
如何將結(jié)構(gòu)數(shù)據(jù)集成到序列搜索中尚不清楚,但一種似乎可行的方法是不直接使用結(jié)構(gòu)數(shù)據(jù),而是通過所謂的嵌入間接使用,它們是由神經(jīng)網(wǎng)絡(luò)生成的中間序列表示,構(gòu)成神經(jīng)網(wǎng)絡(luò)結(jié)構(gòu)預(yù)測(cè)的基礎(chǔ)。
然而,基于嵌入和結(jié)構(gòu)數(shù)據(jù)的同源檢測(cè)只有以易于使用的方式提供并被社區(qū)廣泛采用,才會(huì)有助于改變分子生物學(xué)。NCBI、EBI 和 Riken 等著名機(jī)構(gòu)現(xiàn)在應(yīng)該努力采用 FoldSeek 中實(shí)現(xiàn)的快速結(jié)構(gòu)搜索,或使用嵌入來擴(kuò)展經(jīng)典的基于 BLAST 的蛋白質(zhì)序列搜索,以便 Protein BLAST 繼續(xù)成為未來的趨勢(shì)。
- 免責(zé)聲明
- 本文所包含的觀點(diǎn)僅代表作者個(gè)人看法,不代表新火種的觀點(diǎn)。在新火種上獲取的所有信息均不應(yīng)被視為投資建議。新火種對(duì)本文可能提及或鏈接的任何項(xiàng)目不表示認(rèn)可。 交易和投資涉及高風(fēng)險(xiǎn),讀者在采取與本文內(nèi)容相關(guān)的任何行動(dòng)之前,請(qǐng)務(wù)必進(jìn)行充分的盡職調(diào)查。最終的決策應(yīng)該基于您自己的獨(dú)立判斷。新火種不對(duì)因依賴本文觀點(diǎn)而產(chǎn)生的任何金錢損失負(fù)任何責(zé)任。





 新火種
2023-12-20
新火種
2023-12-20 






