LLM與藥物發現結合,NVIDIA、Mila、Caltech團隊發布多模態分子結構-文本模型

作者|劉圣超
編輯| 凱霞
從2021年開始,大語言和多模態的結合席卷了機器學習科研界。
最近隨著大模型、多模態的應用,一個很自然的想法就是我們是否也可以將這些技術用到藥物發現上?并且這些自然語言的文本描述,是否對于藥物發現這個有挑戰性的問題帶來新的視角?答案是肯定并且樂觀的。
近日,加拿大蒙特利爾學習算法研究院(Mila)、NVIDIA Research、伊利諾伊大學厄巴納-香檳分校(UIUC)、普林斯頓大學和加州理工學院的研究團隊,通過對比學習策略共同學習分子的化學結構和文本描述,提出了一種多模態分子結構-文本模型 MoleculeSTM。
該研究以為題《Multi-modal molecule structure–text model for text-based retrieval and editing》為題,于 2023 年 12 月 18 日發表在《Nature Machine Intelligence》上。
其中劉圣超博士是第一作者,NVIDIA Research 的 Anima Anandkumar 教授為通訊作者。聶維梨、王程鵬、盧家睿、喬卓然、劉玲、唐建和肖超瑋為共同作者。
該項目是劉圣超博士在2022年3月加入 NVIDIA Research之后,在聶維梨老師、唐建老師、肖超瑋老師和 Anima Anandkumar 老師的指導下進行的。
劉圣超博士表示:「我們的動機就是對LLM和藥物發現進行初步的探索,并最終提出了MoleculeSTM。」
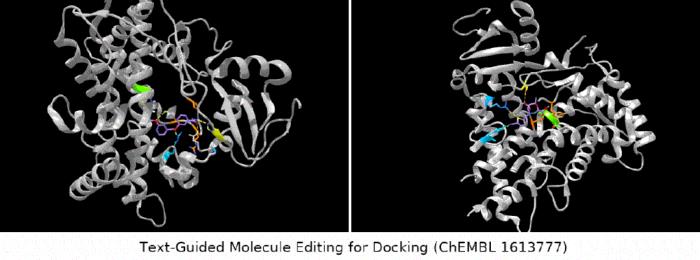
用于對接的文本引導分子編輯。
MoleculeSTM的核心思路非常簡單直接:分子的描述有內部化學結構(internal chemical structure)和外部功能描述(external textual descriptions)兩大類,而我們這里利用了contrastive pretraining的思路,將兩種類型的信息進行alignment聯系。如下圖。
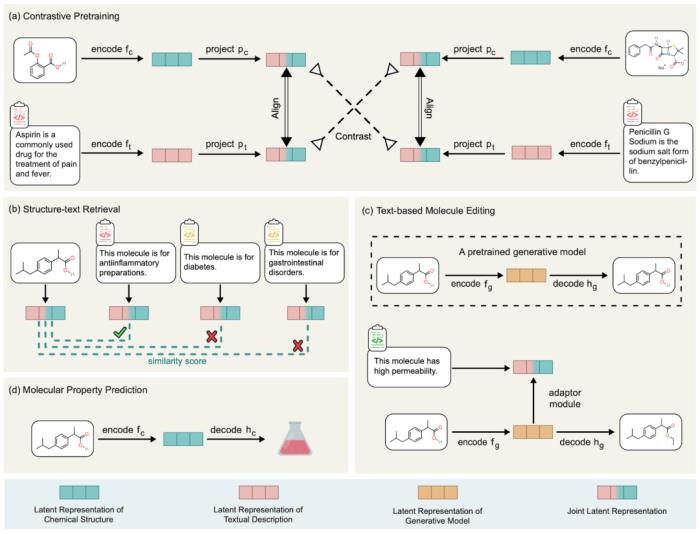
圖示:MoleculeSTM的流程圖。
而MoleculeSTM的這個alignment有一個非常好的性質:當有一些任務在化學空間比較難以解決的時候,我們可以將其transfer到自然語言(natural language)空間。而且自然語言的任務會相對更容易解決,由于它的特性。并且基于此我們設計了種類豐富的下游任務來驗證其有效性。下面我們圍繞幾個insight詳細討論。
自然語言和大語言模型的特性
這個是我們在MoleculeSTM中首先提出的問題。在MoleculeSTM中,我們是利用了自然語言的open vocabulary和compositionality特性:
Open vocabulary的意思是我們可以把現在人類知識都用自然語言表示,所以對于將來新出現的知識,也能用現有的語言進行歸納和總結。比如出現某種新的蛋白質,我們希望可以對它的功能進行自然語言描述。Compositionality的意思是在自然語言中,一個復雜的概念可以用幾個簡單的概念進行聯合表述。這個對于類似多屬性編輯的任務有很大的幫助:在化學空間要編輯分子同時符合多個特性非常困難,但是我們可以非常簡單地用自然語言表達出來多種特性。特性引出的任務設計
現有的language-image task可以認為是藝術相關的任務(比如生成圖片、文字),也就是說它們的結果是可以多樣和不確定。但是科學發現是科學問題,通常有著比較明確的結果,比如生成有某個功效的小分子。這個在任務的設計上帶來了更大的挑戰。
在MoleculeSTM中 (Appendix B),我們提出了兩個準則:
首先我們考慮的任務是能夠進行計算模擬得到結果。將來會考慮能夠有wet-lab驗證的結果,但這并不在目前這個工作的考量范疇內。其次我們只考慮有著模糊性結果的問題。具體例子比如讓某個分子的水溶性或者穿透性變強。而有一些問題有明確結果,比如在分子的某一個位置加入某一個官能團,我們認為這類任務對于藥物、化學專家來說更加簡單直接。所以它可以將來當作某一個proof-of-concept任務,但是并不會成為主要的任務目標。由此我們設計了三個大類任務:
Zero-shot 結構文本檢索;Zero-shot 基于文本的分子編輯;分子性質預測。接下來我們會重點介紹一下第二個任務。
分子編輯的定性結果
這個任務就是同時輸入一個分子和自然語言描述(比如額外的屬性),然后希望能夠輸出復合語言文本描述的新的分子。這就是文本編輯優化(text-guided lead optimization)。
具體的方法就是利用已經訓練好的分子生成模型和我們預訓練好的MoleculeSTM,通過學習二者的潛在空間(latent space)的alignment,從而進行 latent space interpolation,再經過解碼生成目標分子。流程示意圖如下。
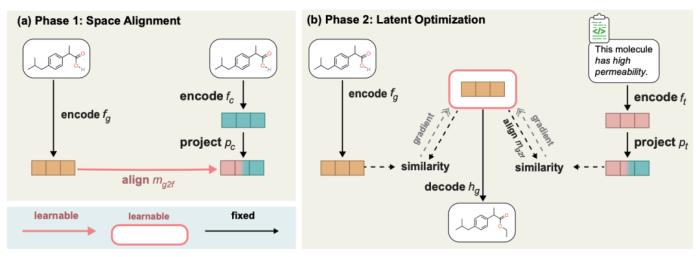
圖示:zero-shot text-guided molecule editing 兩階段流程示意圖。
這里我們展示了幾組分子編輯的定性結果(其余下游任務的結果細節可以參考原論文)。主要我們考慮四類分子編輯任務:
單一屬性編輯:對單一屬性進行編輯,比如水溶性、穿透性、氫鍵施主與受主個數。復合屬性編輯:同時對多個屬性進行編輯,比如水溶性和氫鍵施主個數。藥物相似性編輯:(Appendix D.5)是讓輸入分子與目標分子藥物長得更加接近。專利藥物的鄰居搜索:對于已經申請到專利的藥物,往往會把中間過程的藥物一起報道。我們這里就是那中間藥物配合自然語言描述,看是否能夠生成最終的目標藥物。binding affinity編輯:我們選擇幾個ChEMBL assay作為靶點,目標是讓輸入分子和靶點有更高的結合親和力。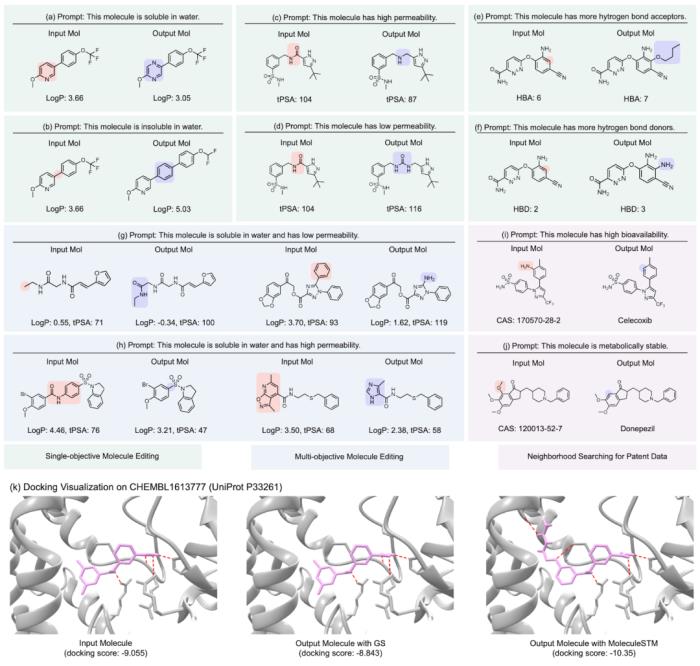
圖示:zero-shot text-guided molecule editing 結果展示。
更有意思的是最后一類任務,我們發現MoleculeSTM的確能夠在緊緊依靠對于靶蛋白的文字描述,而進行配體的配體 先導化合物優化。(注:這里的蛋白質結構信息都是在evaluation是才會知道。)
- 免責聲明
- 本文所包含的觀點僅代表作者個人看法,不代表新火種的觀點。在新火種上獲取的所有信息均不應被視為投資建議。新火種對本文可能提及或鏈接的任何項目不表示認可。 交易和投資涉及高風險,讀者在采取與本文內容相關的任何行動之前,請務必進行充分的盡職調查。最終的決策應該基于您自己的獨立判斷。新火種不對因依賴本文觀點而產生的任何金錢損失負任何責任。





 新火種
2024-01-03
新火種
2024-01-03 






