準確率達95%,混合深度學習搜索納米生物材料,登Nature子刊

編輯 | 蘿卜皮
超分子肽基材料具有革新納米技術和醫學等領域的巨大潛力。然而,破譯其實際應用所必需的復雜序列到組裝途徑仍然是一項艱巨的任務。
它們的發現主要依賴于需要大量資金的經驗方法,這阻礙了它們的顛覆性潛力。因此,盡管自組裝肽種類繁多,且具有明顯的優勢,但只有少數肽材料進入了市場。
基于實驗驗證數據進行訓練的機器學習可以快速識別具有高自組裝傾向的序列,從而將資源支出集中在最有前途的候選序列上。
克羅地亞里耶卡大學 (University of Rijeka)的研究人員介紹了一個框架,該框架在基于元啟發式的生成模型中實現了精確的分類器,以在具有挑戰性的肽序列空間中進行搜索。
為此,該團隊訓練了五個循環神經網絡,其中使用聚集傾向和特定物理化學性質的序列信息的混合模型取得了優異的性能,準確率為 81.9%,F1 得分為 0.865。
分子動力學模擬和實驗驗證已證實,生成模型在自組裝肽的發現中準確率為 80-95%,優于目前最先進的模型。
該研究以「Reshaping the discovery of self-assembling peptides with generative AI guided by hybrid deep learning」為題,于 2024 年 11 月 19 日發布在《Nature Machine Intelligence》。

分子自組裝(SA)是由非共價弱相互作用驅動的基本化學過程,肽作為結構多樣的分子構件,能夠組裝成復雜的超分子材料。然而,實驗發現新型自組裝肽效率低、成本高,并因搜索空間龐大而受限。
分子動力學(MD)模擬和機器學習(ML)為肽設計提供了新思路,尤其是ML模型通過更快的運算和更高效的數據處理,展現了在肽自組裝預測中的潛力。
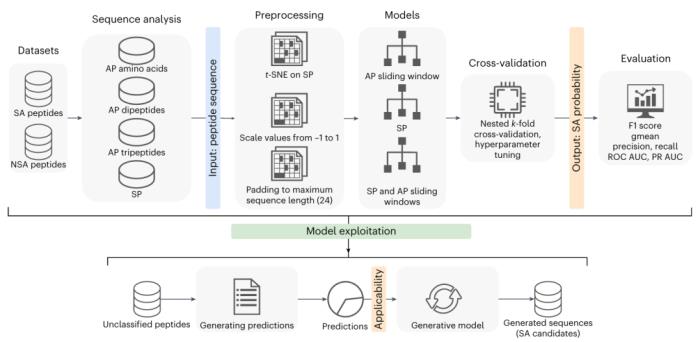
圖示:擬議研究流程的概述。(來源:論文)
在最新的研究中,里耶卡大學的研究團隊開發了一種基于 RNN 的方法,使用不規則采樣的不等長特征來評估未分類肽的 SA 潛力,該方法基于氨基酸、二肽和三肽的 AP 分數作為任何給定目標肽的預測變量。
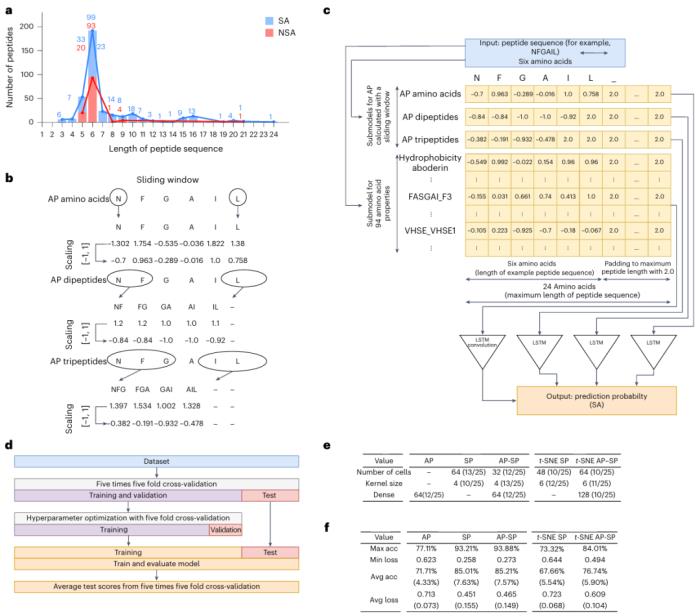
圖示:從數據集到滑動窗口機制和超參數優化的神經網絡設置。(來源:論文)
此外,RNN 分類器用作基于搜索的遺傳算法中的適應度函數,以形成生成模型,用于發現具有高 SA 傾向的序列。該模型補充了人類的直覺,試圖基于 ML 輔助的無偏序列空間探索來識別具有高 SA 傾向的新肽。
具體來說,研究人員通過改變架構、輸入數據和訓練參數,開發了五種基于序列到組裝 RNN 的預測模型。使用通過長度為 1、2 或 3 個殘基的滑動窗口獲得的預先計算的 AP 分數以及特定的物理化學特性,然后用從文獻中整理的實驗數據對模型進行訓練。這使得模型能夠分析任意長度的序列,而無需使用 MD 進行大量的 AP 分數計算。
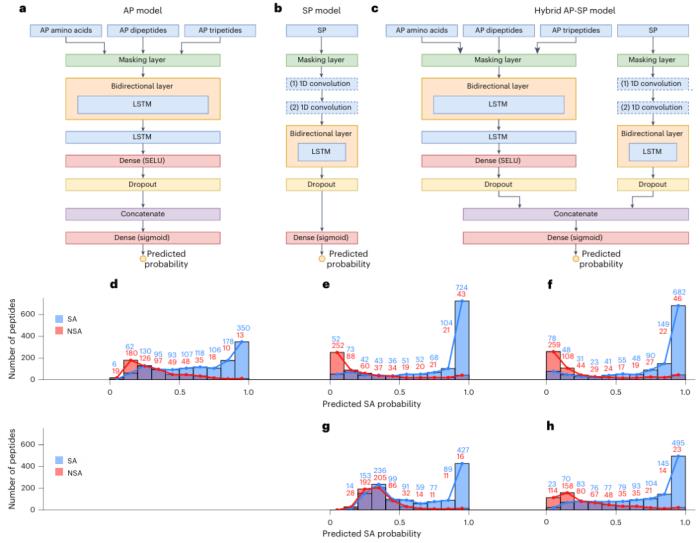
圖示:RNN 架構和各種性能評估。(來源:論文)
混合 AP–SP 模型可區分 SA 和 NSA 肽,F1 得分高達 0.865,并且其將知識推廣到現有數據集未探索的化學空間區域的能力已在生成模型中進行測試。使用 MD 模擬對生成的肽(十個 SA 和十個 NSA)進行驗證,證實了模型精度為 90–100%。
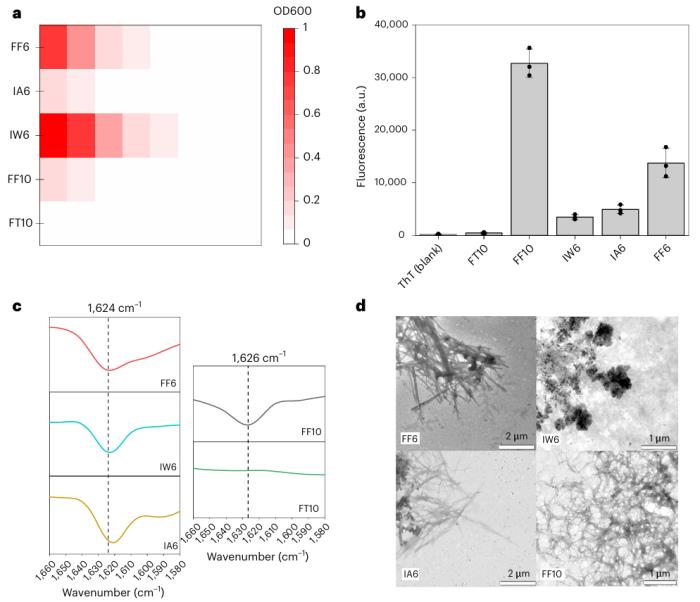
研究人員對三種六肽和兩種十肽進行了真實實驗驗證。OD、衰減全反射 (ATR)-FTIR、ThT測定和 TEM 測量證實,五種肽中有四種發生自組裝,這與 ML 引導生成模型中使用的 AP-SP 分類器 (81.9%) 的準確率一致。
因此,生成模型的表現優于人類和人工智能專家,準確率高出 25% 至 35%。鑒于現有 SA 推理方法的資源密集型特性,ML 模型可以精確定位具有 SA 高度傾向的序列,同時所需的時間和資源更少。
研究人員相信,生成模型的準確性表明,他們開發的 ML 模型成功捕獲了存儲在實驗驗證數據中的底層規則。這在發現具有高自組裝概率的肽方面,提供了一種補充人類直覺的方法,并為未來智能和自動駕駛實驗室的發展提供了機會,從而可以更快、更可持續地發現新材料。
論文鏈接:
- 免責聲明
- 本文所包含的觀點僅代表作者個人看法,不代表新火種的觀點。在新火種上獲取的所有信息均不應被視為投資建議。新火種對本文可能提及或鏈接的任何項目不表示認可。 交易和投資涉及高風險,讀者在采取與本文內容相關的任何行動之前,請務必進行充分的盡職調查。最終的決策應該基于您自己的獨立判斷。新火種不對因依賴本文觀點而產生的任何金錢損失負任何責任。





 新火種
2024-12-13
新火種
2024-12-13 






