穩健且準確,AlphaFold結合兩種AI方法,實現蛋白質化學位移自動分配
編輯 | 綠蘿
化學位移分配對于基于核磁共振 (NMR) 的蛋白質結構、動力學和相互作用研究至關重要,可提供重要的原子級見解。然而,獲得化學位移分配是勞動密集型的并且需要大量的測量時間。
為了解決這一限制,蘇黎世聯邦理工學院(ETH Zurich)的研究人員之前提出了 ARTINA——一種用于自動分配二維 (2D)–4D NMR 譜的深度學習方法。
近日,研究人員提出了一種將 ARTINA 與 AlphaFold 和 UCBShift 相結合的綜合方法,能夠減少實驗數據、提高準確性并增強大型系統的穩健性,從而實現化學位移分配。
研究證明,與使用所有實驗可用的 NMR 數據(平均每個蛋白質 10 個 3D 光譜,91.37%)的純 ARTINA 運行相比,五個 3D 光譜產生更準確的分配(92.59%),從而大大減少了所需的測量時間。研究還報告了在多達 500 個殘基的大型合成系統中提高了分配準確性。
該研究以《Time-optimized protein NMR assignment with an integrative deep learning approach using AlphaFold and chemical shift prediction》為題,于 2023 年 11 月 22 日發布在《Science Advances》上。
核磁共振 (NMR) 光譜是一項關鍵的分析技術,可提供蛋白質結構、動力學和相互作用的詳細信息。
使用本質上存在的核自旋探針可以同時獲得大量單個原子位置的這些數據。為了實現這種原子分辨率,必須將核自旋的共振頻率(以化學位移表示)歸因于蛋白質中的各個原子。這種化學位移分配是大多數蛋白質 NMR 研究的一項關鍵任務。尋找化學位移分配非常耗時并且需要專業知識。
為了加速 NMR 化學位移分配來改變這種情況,人們應該減少所需光譜的數量,并在不影響結果可靠性的情況下實現分析的自動化。
在此,研究人員提出了一種方法,通過利用機器學習的最新進展并將三維(3D)蛋白質結構中包含的信息有效地納入分配過程來實現這一目標。后者用于取代必須從額外的核磁共振譜中收集的信息。
通用自動分配方法 FLYA 最近被嵌入到 NMR 應用人工智能方法 (ARTINA) 和 NMRtist 網絡服務器中,它們使用機器學習進行可視化光譜分析和其他任務,以自動完成從原始光譜到分配和 3D 結構的蛋白質核磁共振數據分析的整個過程。ARTINA 基本上減少了蛋白質化學位移分配的工作量,減少了樣品制備和光譜測量的工作量。
然而,平均而言,需要使用超過 13 個多維 NMR 譜才能獲得這些結果,這相當于使用傳統采集方案對每個蛋白質進行 2 周以上的 NMR 測量時間。考慮到 ARTINA 算法的計算時間通常小于 2 小時,并且 NMR 波譜儀的操作比計算機的操作成本更高且要求更高,因此減少用于分配的光譜數量是提高生物分子核磁共振項目效率的明顯策略。
蛋白質 3D 結構的知識主要可以通過兩種方式支持化學位移分配:通過更真實地預測核歐佛豪瑟效應頻譜(NOESY)中的預期交叉峰,以及通過基于結構的化學位移值的預測。這變得尤為重要,因為 AlphaFold 現在可以準確預測大多數結構化蛋白質的 3D 結構。
ARTINA 與 AlphaFold 和 UCBShift 相結合的綜合方法基于此,研究人員建立了一種基于機器學習的綜合方法,用于基于結構的 NMR 化學位移分配。評估了其性能并確定用于分配主鏈酰胺基團或所有化學位移的最佳光譜集。
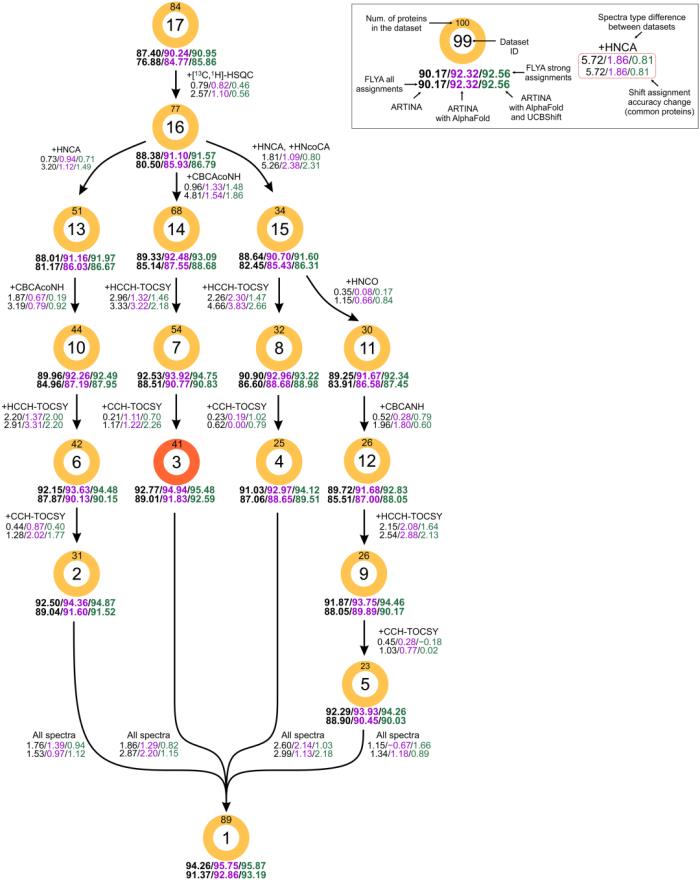
從 ARTINA 原始出版物的 89 種蛋白質的 1170 個實驗光譜開始,研究定義了 25 組不同的輸入光譜,用于 ARTINA 的自動分配計算。
化學位移分配的準確性在三種不同的情況下進行了評估:通過「經典」三共振譜對主鏈酰胺基團進行分配,通過 3D NOESY 和三共振譜對主鏈酰胺基團進行分配,以及完成主鏈和側鏈化學位移的分配,并在每種情況下比較 ARTINA 在沒有結構輸入的情況下獲得的分配結果,或僅使用結構生成預期的 NOESY 交叉峰,或者使用 UCBShift(一種化學位移計算程序)方法將該結構用于預測化學位移。
此外,研究使用僅用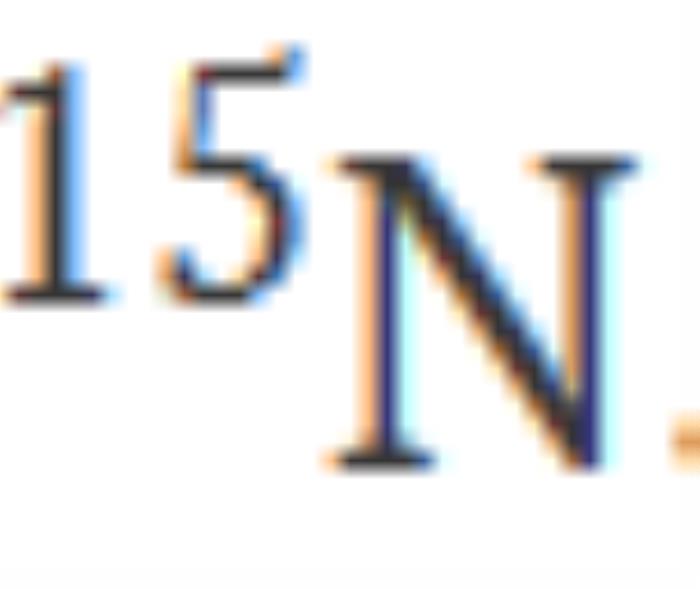 標記樣品記錄的 NMR 譜測試了主鏈酰胺分配的可行性,這可以構成昂貴的
標記樣品記錄的 NMR 譜測試了主鏈酰胺分配的可行性,這可以構成昂貴的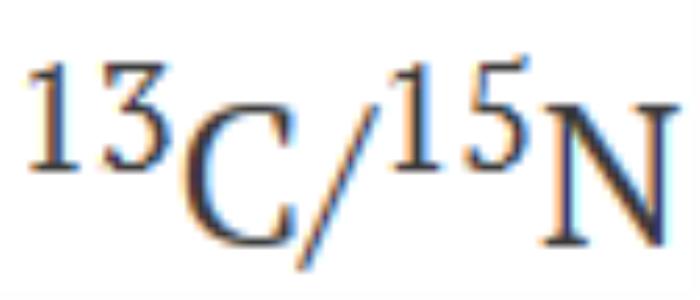 雙標記的替代方案。
雙標記的替代方案。
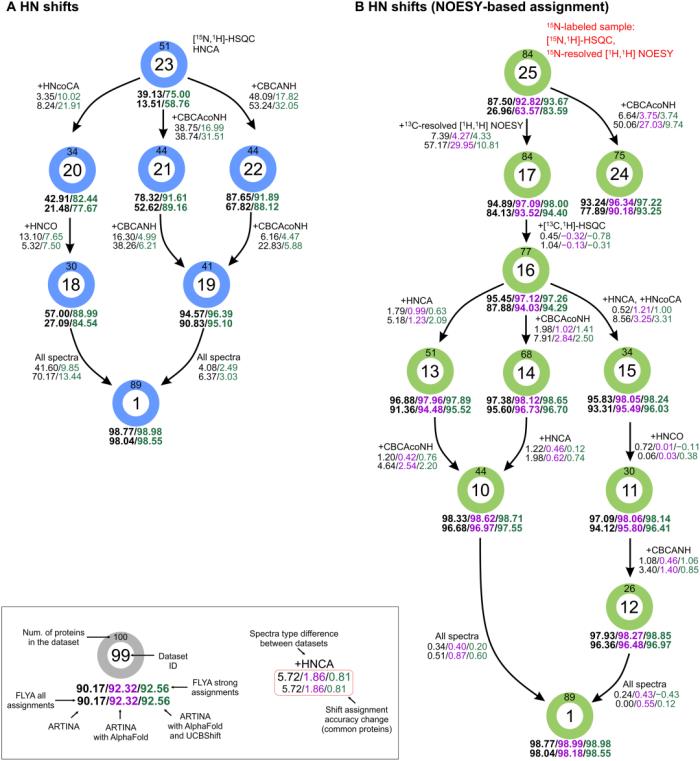
圖 2:實驗數據量對主鏈酰胺化學位移分配準確性的影響。(來源:論文)
研究人員使用 3DRobot 生成的大量折疊良好且包裝良好的 decoys 來評估輸入結構的準確性對分配的影響,這些 decoys 與實驗結構的偏差為 0 到 5? 均方根偏差(RMSD)。
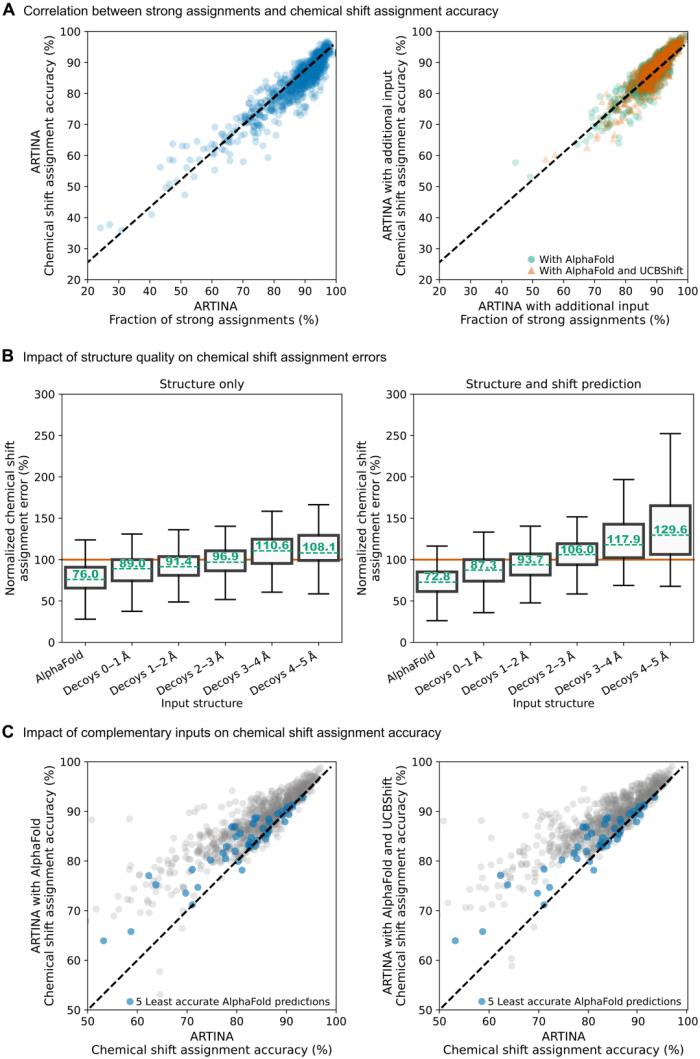
研究人員還評估了綜合方法如何分配大型合成生成的蛋白質系統(最多 500 個殘基),這些系統目前很少存放在生物磁共振數據庫 (BMRB) 數據庫中。
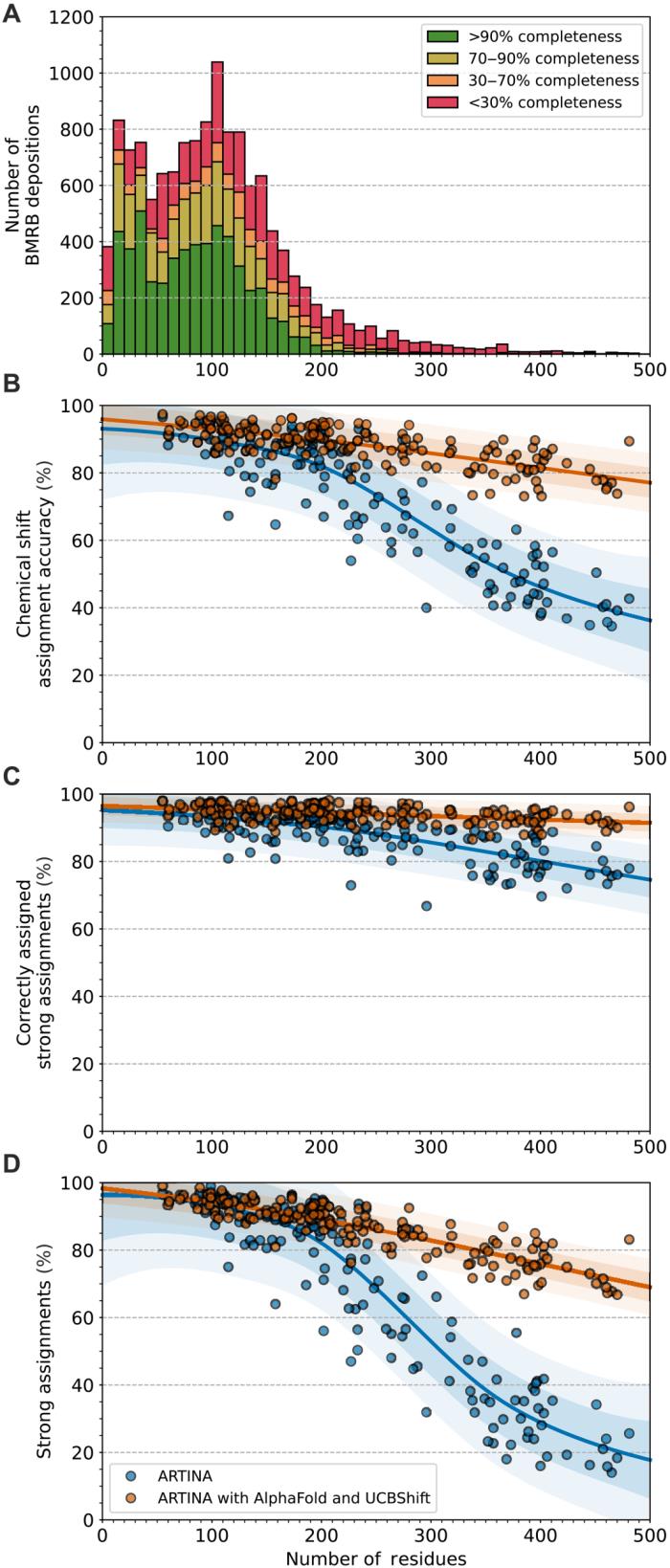
總之,結合該綜合方法及其評估,研究人員提出了一套數據驅動的實用建議,用于執行蛋白質的化學位移分配。
綜合方法還具有研究其他類型系統的潛力,例如蛋白質復合物、固有無序蛋白質、膜蛋白以及細胞內和固態核磁共振,對于這些系統,機器學習方法與單體系統同樣有前景。
- 免責聲明
- 本文所包含的觀點僅代表作者個人看法,不代表新火種的觀點。在新火種上獲取的所有信息均不應被視為投資建議。新火種對本文可能提及或鏈接的任何項目不表示認可。 交易和投資涉及高風險,讀者在采取與本文內容相關的任何行動之前,請務必進行充分的盡職調查。最終的決策應該基于您自己的獨立判斷。新火種不對因依賴本文觀點而產生的任何金錢損失負任何責任。





 新火種
2023-12-01
新火種
2023-12-01