分子對接速度提升10,000倍,深度圖學習加速RNA虛擬篩選,助力藥物靶標發現
編輯 | 蘿卜皮
RNA 是尚未開發的藥物靶標的巨大寶庫。基于結構的虛擬篩選 (VS) 利用結合位點信息識別候選分子,傳統上采用分子對接模擬。然而,對接很難在大型化合物庫和 RNA 靶標中擴展。
機器學習提供了一種解決方案,但由于數據和實際評估有限,它在 RNA 方面的開發仍不夠充分。
麥吉爾大學(McGill University)、馬克斯普朗克生物化學研究所(Max Planck Institute of Biochemistry)、巴黎綜合理工學院(Ecole Polytechnique)的研究人員提出了針對 RNA 量身定制的數據驅動 VS 流程,利用粗粒度 3D 建模、合成數據增強和 RNA 特定的自我監督。
該模型實現了對接速度的 10,000 倍提升,同時在結構不同的測試集上將活性化合物排在前 2.8%。它對結合位點變異具有很強的穩健性,并成功地從 20,000 種化合物體外微陣列中篩選出未知的 RNA 核糖開關,平均富集因子為 2.93,1% 。這標志著基于結構的 RNA VS 深度學習首次通過實驗驗證獲得成功。
該研究以「RNAmigos2: accelerated structure-based RNA virtual screening with deep graph learning」為題,于 2025 年 3 月 21 日發布在《Nature Communications》。

只有一小部分 RNA 編碼蛋白質,而 ncRNA 目前已知在廣泛的生物過程中發揮著關鍵作用。例如,大約 2000 個基因編碼 micro-RNA,而 micro-RNA 又影響 60% 基因的表達。盡管 micro-RNA 無處不在,但第一種 RNA 靶向藥物 risdipalm 直到最近才獲得 FDA 批準,而且幾乎所有市售的小分子療法仍然以蛋白質為靶點。
靶向 RNA 的能力將大幅增加可用藥物的空間,并在過度使用的蛋白質靶點不足的情況下提供替代方案。例如,lncRNA 可以代表腫瘤學中有趣的治療靶點,而蛋白質靶點可能過于專業化。RNA 靶標也代表了治療缺乏蛋白質靶標的疾病(如三陰性乳腺癌)的一種途徑。
在此背景下,RNA 越來越被認為是開發新型小分子療法的有前途的靶標家族,這凸顯了對 RNA 藥物發現的有效工具的需求。
在最新的研究中,麥吉爾大學等機構的研究人員提出了一種基于結構的 RNA 虛擬篩選方法——RNAmigos2,該方法與分子對接相比只需極短的時間,為大規模基于靶標的 RNA 藥物發現打開了大門。
RNAmigos2 旨在使用查詢 RNA 結構快速篩選配體庫中的結合物。其工作流程如下圖所示,以候選結合位點結構(作為完整 3D 或堿基配對網絡)和要篩選的化合物列表作為輸入。然后,該工具會為每種化合物返回一個反映結合可能性的分數。
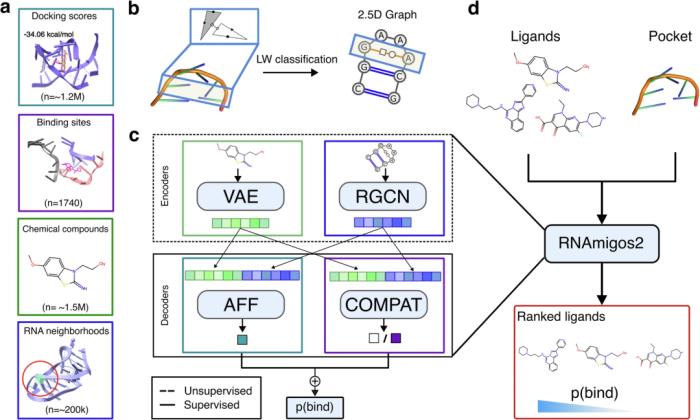
圖示:RNAmigos2 化合物篩選流程概述。(來源:論文)
RNAmigos2 架構
RNAmigos2 模型采用編碼器-解碼器框架,具有兩個編碼器和兩個解碼器,每個編碼器和解碼器都在不同的數據源上進行訓練。兩個編碼器分別將輸入的 RNA 結合位點和小分子映射到嵌入中。RNA 3D 結構表示為一個稱為 2.5D 圖的圖,該圖對結構中發生的所有規范(Watson-Crick 和 Wobble)和非規范堿基對相互作用進行編碼。
這種表示使研究人員能夠用適合機器學習框架的離散數學對象捕捉 RNA 3D 結構的關鍵特征,并且被證明是 RNA 化學信息學應用的有用生物學先驗。
RNA 編碼器以 2.5D 圖作為輸入,并學習使用自監督訓練方案在所有可用的非冗余 RNA 子結構上生成 RNA 表征。配體以分子圖表示。配體編碼器使用在 中提出并在大量化合物數據集上訓練的變分自動編碼器模型來學習配體的神經表征。
為了訓練解碼器,研究人員從 PDB 中提取了 1740 個 RNA-配體復合物,并將它們分組到 436 個相似的結合位點簇中,他們使用 RMAlign 以 0.75 的相似性閾值識別了這些結合位點。這種方法代表了對 RNA 藥物靶標關聯預測的嚴格基于結構的劃分。
第一個解碼器 (Compat) 被訓練為二元分類器,以區分結合位點的天然配體和誘餌。此外,為了綜合增加 PDB 化合物的有限數量和藥物相似性,研究人員進行了大規模對接實驗,將 500 種藥物類 ChEMBl 化合物對接在 1740 個結合位點上。
第二個解碼器經過訓練后,可以使用對接數據預測結合親和力 (Aff)。給定一個結合位點和配體列表,研究人員對所有對象進行編碼,并使用聯合解碼器預測可用于虛擬篩選的兼容性分數。之后,該團隊通過模型為活性化合物分配高分的能力來衡量模型的性能,而不是為非活性(誘餌)化合物池分配高分。
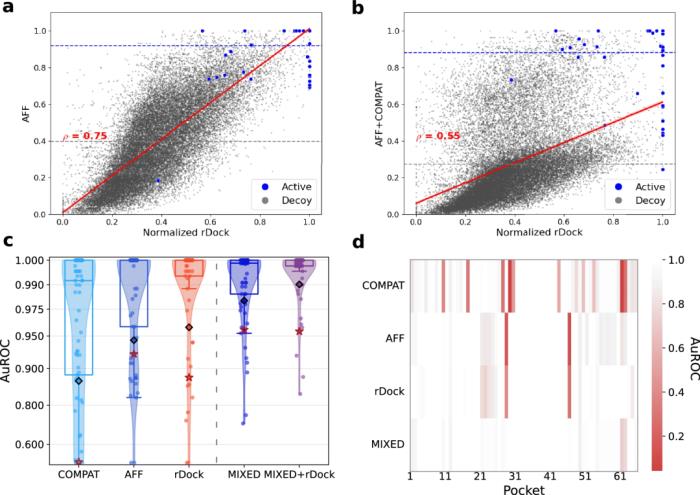
圖示:RNAmigos2 模型集成基準。(來源:論文)
性能強悍
測試顯示,該模型運行速度比對接快一萬倍以上。盡管運行時間僅需數秒而非數小時,但所提出的方法可檢索到更高的分子對接富集因子(候選配體列表的前 2.8% vs 4.1%)。
這一結果對口袋身份相對敏感,但對口袋擾動足夠穩健,可與現代口袋挖掘算法結合使用。此外,通過將模型與得分最高的化合物的實際對接分數相結合,研究人員設法將對接錯誤率降低了四倍,所用時間減少了四分之一。
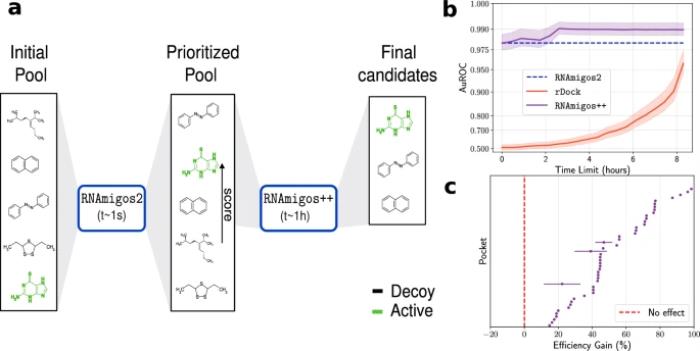
圖示:RNAmigos2 輔助的虛擬篩選效率。(來源:論文)
團隊在獨立的大規模(25k 種化合物)體外結合篩選中確定了該工具的性能,并表明它在兩個 CPU 核心分鐘內提供了 2.93 的 1% 的富集因子。這些結果共同確立了 RNAmigos2 作為基于結構的 RNA 虛擬篩選的最新水平。研究人員已經公開發布了他們所有的數據集、源代碼和模型權重,希望激發社區朝著這個重要方向努力。
相關代碼:
https://doi.org/10.5281/zenodo.14803961
https://github.com/cgoliver/rnamigos2/
目前,該方法的局限性包括需要預定義的結合位點,為此需要開發與結合位點預測器的集成,以及對結合位點靈活性進行建模。
未來研究的一個有趣方向是研究使用其他對接工具來訓練其他模型,從而產生快速替代模型,這些替代模型可能因不同的評分函數而產生不同的錯誤模式。
研究人員設想,RNAmigos2 等工具將與迅速興起的以 RNA 為中心的分子設計技術和新發布的支持核酸的 AlphaFold3 發揮協同作用,為下一代 RNA 藥物發現鋪平道路。
值得注意的是,該方法具有獨特的優勢,只需手頭有低分辨率結構數據(例如堿基對相互作用)即可實現基于結構的 RNA 虛擬篩選。鑒于潛在的 RNA 靶標數量驚人,這一特性可能成為挖掘整個基因組并全面迎接 RNA 治療時代的重要資產。
論文鏈接:https://www.nature.com/articles/s41467-025-57852-0
- 免責聲明
- 本文所包含的觀點僅代表作者個人看法,不代表新火種的觀點。在新火種上獲取的所有信息均不應被視為投資建議。新火種對本文可能提及或鏈接的任何項目不表示認可。 交易和投資涉及高風險,讀者在采取與本文內容相關的任何行動之前,請務必進行充分的盡職調查。最終的決策應該基于您自己的獨立判斷。新火種不對因依賴本文觀點而產生的任何金錢損失負任何責任。





 新火種
2025-03-26
新火種
2025-03-26