Nature子刊新登,如何檢測小分子機器學習中的覆蓋率偏差

編輯丨&
小分子機器學習旨在從分子結構中預測化學或生物特性,其應用包括毒性預測、配體結合和藥代動力學等。
最近的一個趨勢是開發避免顯式領域知識的端到端模型。這些模型假設訓練和評估數據中沒有覆蓋率偏差,這意味著數據代表了真實分布。
來自德國的一支聯合研究團隊探索了大規模數據集對已知生物分子結構空間的覆蓋程度,并提出了一種基于求解最大公共邊子圖(MCES)問題的距離度量,該問題與化學相似性非常吻合。
他們的研究結果以「Coverage bias in small molecule machine learning」為題,于 2025 年 1 月 9 日刊登于《Nature Communications》。

研究結果表明,許多廣泛使用的數據集缺乏生物分子結構的統一覆蓋,這限制了在其上訓練的模型的預測能力。為此,他們提出了兩種額外的方法來評估訓練數據集是否與已知的分子分布不同,從而可能指導未來的數據集創建以提高模型性能。
數據集的介紹
幾十年來,機器學習已成功應用于生物化學和化學領域。最近的趨勢是開發端到端模型,避免通過歸納偏差顯式整合領域知識。不應在其適用范圍之外使用模型這一事實在化學計量學界已經廣為人知。在空間偏差中,人們使用來自某個地理位置的測試數據,但也對模型在其他位置的性能做出聲明。
而在訓練用于預測分子特性的大規模端到端模型時,這個問題通常會被忽略。目前,數據集內的泛化問題已得到廣泛研究。對于小分子,廣泛使用的支架分割可確保對訓練數據中未看到的支架進行評估。雖然這么做并不能解釋分子性質分布的差異。
要考慮小分子的訓練數據分布,需要某種方法來估計分子結構之間的相似性或差異性。雖然可以采用分子指紋以快速處理大型數據集,但是基于分子指紋的測量會表現出不良特征。而基于最大公共子圖的方法雖然可以更好的捕捉結構性相似的化學直覺,但是計算過程過于麻煩。
團隊展示了如何檢查分子結構數據集是否覆蓋了生物學感興趣的小分子結構。自然產物相似性的度量可以很好地指示數據集中分子結構的分布是否與生物分子結構的分布有很大差異。
人類目前尚未真正了解具有生物學意義的小分子宇宙,團隊提出了這點,因為人類還有未曾發現的小分子。雖然目前作為代理的生物分子結構庫還不夠完整,但是對適用域的限制已經顯現出來。
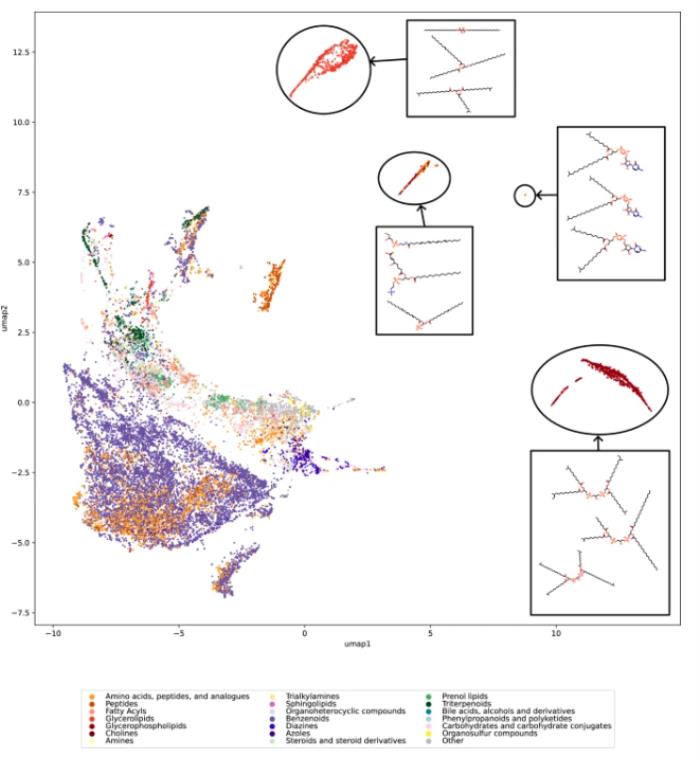
對于給定的一對分子結構,團隊采用最大公共邊子圖計算了距離。除此之外,他們還估計了所有距離的下限以加快計算速度。均勻流形近似和投影(UMAP)在他們繪制可視化生物分子結構的世界的二維圖里被使用。
為了避免運行時間的增加和雜亂的繪圖,研究團隊對 20,000 個生物分子結構進行了統一二次抽樣。他們觀察到,子采樣確實可能會改變 UMAP 嵌入的一般布局,但一般布局通常出奇地相似。
某些分子結構和化合物類別,特別是某些脂質類別,會導致 UMAP 嵌入中出現異常值簇。團隊表示必須要高度謹慎地從 2 維 UMAP 嵌入中推斷數據的結構。
在公共數據集中,團隊觀察到,可用的分子結構子集通常遠非統一。他們認為,大多數公共數據集也不具有代表性,這意味著數據集中完全缺少大面積的生物分子結構。實際上,一些數據集集中在圖中的一個或幾個區域。
對實例進行驗證
團隊考慮了 10 個經常用于訓練機器學習模型的公共分子結構數據集。他們研究了每個數據集中的分子結構在多大程度上是生物分子結構的統一子集,并算了所有分子結構的近視 MCES 距離。
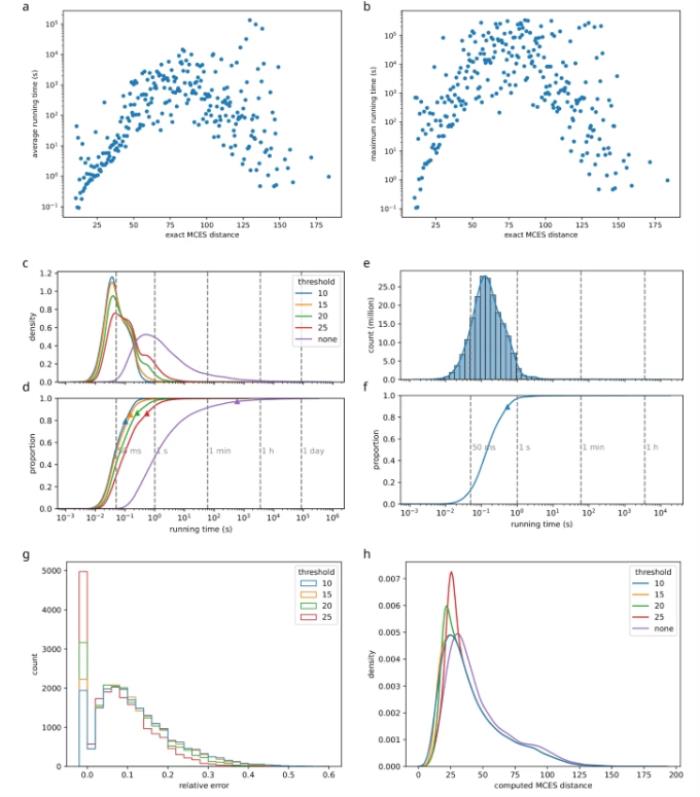
首先考慮 ILP 運行時間對確切 MCES 距離的依賴性,其次評估邊界和精確計算的組合如何產生有利的運行時間。為了通過子采樣排除偏差,團隊使用 19,994 個生物分子結構中的所有對重復了上述分析。最后對計算邊界的誤差進行分析。
與設計的實現類似,計算邊界的結果可用于在未執行精確計算時近似相似性。為了準確計算更多實例,相似性閾值被降低到 0.5。
除了均勻的子樣本外,分子結構數據集還應表現出另一個特征,以便它代表生物分子結構的整個空間:即生物分子所屬的所有化合物類別也應存在于訓練數據中。
如果數據集完全遺漏了特定化合物類別的分子結構,那么根據數據訓練的機器學習模型可能會顯示對該化合物類別的預測不佳。如果特定化合物類的樣本非常少,則情況也是如此。
在研究中,團隊專注于生物分子結構的機器學習模型。因此,他們想忽略基本上不包含生物分子結構的化合物類別。如果沒有或很少有生物分子屬于某個化合物類別,那么分子結構數據集也不包含該化合物類別的分子結構或僅包含少量分子結構也就不足為奇。
UMAP 嵌入引入了一定程度的任意性,允許在沒有化合物類的束縛的情況下發現問題。相比之下,化合物類分析無法檢測訓練數據的所有缺點。
潛在陷阱與改進
包含分子結構實驗數據的機器學習數據集通常與生物分子結構的統一子集有很大不同。更令人擔憂的是,對于大多數數據集來說,生物分子結構宇宙的大部分區域仍然是完全空白的。
由于機器學習在這些領域的重要性日益增加,因此他們發布了幾項關于化學和生命科學領域良好機器學習實踐的指南。對于在小分子上訓練的大型模型,建議將訓練數據的分布分析納入這些建議中。否則,使用更復雜的機器學習模型進行性能改進在實踐中可能不會有任何有效結果。
即使數據集沒有顯示任何特性,但這也并不意味著機器學習可以全權委托。他們所采用的方法可能發現分子結構分布奇特且具有潛在危險的數據集。根據以往的經驗,他們警告讀者,即使在這里,分子結構的分布也可能導致經過訓練的模型出現意外行為和違反直覺的評估結果。
團隊推測,MCES 邊界的 C++ 實現可以達到與 RDKit 的 RASCAL 實現相當的運行時間,特別是 MCES 距離可以用作機器學習的一部分。可以借助它測量分子結構之間的絕對距離,也可以對其進行修改以考慮子結構關系。
原文鏈接:https://www.nature.com/articles/s41467-024-55462-w
- 免責聲明
- 本文所包含的觀點僅代表作者個人看法,不代表新火種的觀點。在新火種上獲取的所有信息均不應被視為投資建議。新火種對本文可能提及或鏈接的任何項目不表示認可。 交易和投資涉及高風險,讀者在采取與本文內容相關的任何行動之前,請務必進行充分的盡職調查。最終的決策應該基于您自己的獨立判斷。新火種不對因依賴本文觀點而產生的任何金錢損失負任何責任。





 新火種
2025-02-13
新火種
2025-02-13