九大成像模式一鍵解析,生物醫學圖像AI再迎突破!微軟、UW等BiomedParse登Nature子刊
作者 |BiomedParse團隊
編輯 | ScienceAI
生物醫學圖像解析在癌癥診斷、免疫治療和疾病進展監測中至關重要。然而,不同的成像模式(如MRI、CT和病理學)通常需要單獨的模型,造成資源浪費和效率低下,未能充分利用模式間的共性知識。
微軟團隊最新發布的基礎模型BiomedParse,開創性地通過文本驅動圖像解析將九種成像模式整合于一個統一的模型中,通過聯合預訓練處理對象識別、檢測與分割任務,實現了生物醫學圖像解析的新突破。
BiomedParse顯著提升了復雜、不規則形狀對象的識別精度,同時降低了用戶交互的需求,為精準醫療和生物醫學發現提供了更強大的工具。

論文鏈接:https://www.nature.com/articles/s41592-024-02499-w
BiomedParse:通過語言打破九種成像模式之間的壁壘
醫學圖像的成像模式差異巨大(如CT、MRI、病理切片、顯微鏡圖像等),傳統上需要訓練專家模型進行處理。然而不同醫學圖像呈現的物體背后,實際上是共通的生物醫學知識。
BiomedParse是第一個通過醫學語言實現跨九種成像模式進行一致性分析的生物醫學基礎模型。
用戶只需通過簡單的臨床語言提示指定目標對象,例如「腫瘤邊界」或「免疫細胞」,BiomedParse便能準確識別、檢測并分割圖像中的相關區域。相比傳統需要手動框定或標注對象邊界的模型,
BiomedParse極大地減少了科學家和臨床醫生的工作量。無論是影像級別的器官掃描,還是細胞級別的顯微鏡圖像,BiomedParse都可以直接利用臨床術語進行跨模式操作,為用戶提供了更統一、更智能的多模式圖像解析方案。
這種跨模式的一體化方法,連接了放射學、病理學、顯微鏡學等多個領域,幫助研究人員從不同模式的數據中解析出有價值的信息,從而探索多尺度、跨學科的生物醫學問題。BiomedParse的問世,標志著生物醫學圖像分析從單一模式走向了全局統一的新階段。
核心亮點
BiomedParse在生物醫學圖像分析中解鎖了多項創新功能:
跨模式一致性分析:BiomedParse首次實現了跨九種成像模式的穩定表現,取代了傳統的單獨工具,使研究人員能夠更快速、便捷地分析大量數據集。文本驅動的圖像解析:BiomedParse利用自然語言提示進行圖像解析,將對象識別、檢測和分割任務視為一體。無需耗時的手動標注或邊界框操作,顯著縮短了大規模圖像分析的時間和精力。復雜結構的精準識別:在分割不規則形狀的生物醫學對象方面,BiomedParse相較傳統模型表現卓越。通過將圖像區域與臨床概念關聯,分割精度比之前最好方法提高了39.6%,確保了在關鍵任務中的可靠性。GPT-4驅動的大規模數據合成
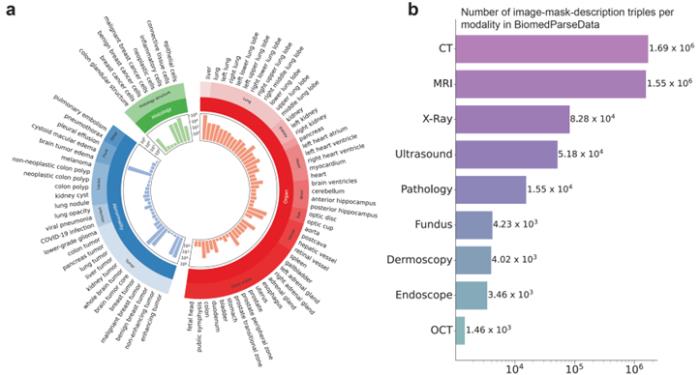
為支持BiomedParse的預訓練,微軟團隊利用GPT-4從45個公開的醫學圖像分割數據集生成了首個覆蓋對象識別、檢測和分割任務的數據集BiomedParseData。
該數據集包含超過600萬個圖像、分割標注與文字描述三元組,涵蓋64種主要生物醫學對象類型及82個細分類別,涉及CT、MRI、病理切片等九種成像模式。通過GPT-4的自然語言生成能力,研究人員將散落在各種現有數據集中的分割任務用統一的醫學概念和語言描述整合起來,讓BiomedParse能在更大,更多樣的數據中融會貫通。
實驗評估:只需文字提示,BiomedParse精度超越SOTA
在測試集上,BiomedParse在Dice系數上顯著超越了當前最優方法MedSAM和SAM,并且無需對每個對象手動提供邊界框提示。即使在給MedSAM和SAM提供精準邊界框的情況下,BiomedParse的純文本提示分割性能仍能超越5-15個百分點。
此外,BiomedParse的性能還優于SEEM、SegVol、SAT、CellViT、Swin UNETR等多個模型,尤其在復雜不規則的對象識別上表現突出。
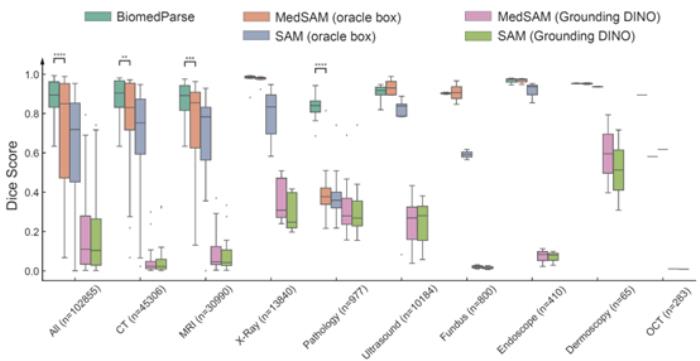
生物醫學圖像中的不規則對象一直是傳統模型的難題,而BiomedParse通過聯合對象識別和檢測任務,通過文本理解實現了對對象特定形狀的建模。對復雜對象的識別精度遠超傳統模型,且在多模態數據集中進一步凸顯了其優勢。
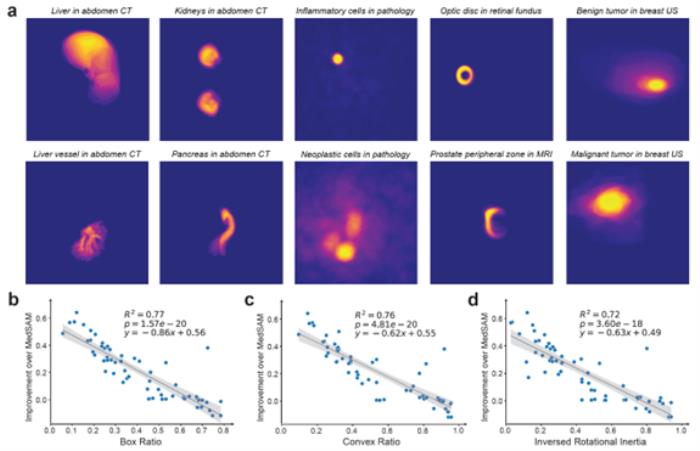
展望未來:多模態生物醫學AI的基石
BiomedParse拓展了生物醫學圖像解析的可能性,將九種成像模式納入一個統一、多用途的模型中。通過簡單的文本提示,BiomedParse顯著減少了用戶的交互需求,尤其在包含大量對象的圖像中,無需逐一標注對象的邊界框。通過對象識別閾值建模,BiomedParse能夠檢測無效的提示請求,并在圖像中不存在指定對象時拒絕分割。
BiomedParse可以一次性識別并分割圖像中的所有已知對象,實現全局圖像解析的擴展,未來有望應用于早期檢測、預后評估、治療決策支持和疾病進展監測等精準醫療關鍵應用場景。
展望未來,BiomedParse擁有廣闊的發展潛力,可進一步擴展至更多成像模式和對象類型,并與LLaVA-Med等高級多模態框架集成,支持「對話式」圖像分析,實現數據交互式探索。
為促進生物醫學圖像分析研究,微軟已將BiomedParse開源并提供Apache 2.0許可,相關演示demo 和 Azure API均已上線,以支持全球精準醫療和健康研究的進步。
微軟布局醫療AI
近年來,微軟在醫療人工智能(AI)領域積極布局,取得了多項重要成果。今年初,微軟聯合華盛頓大學和Providence醫療系統發布了首個全切片數字病理學模型GigaPath,該研究成果發表在《自然》正刊上。
近期,微軟在其Azure AI平臺上部署了多個多模態醫療AI模型,包括本文中提到的BiomedParse和專為放射學應用設計的生成式多模態AI模型LLaVA-Rad和MAIRA-2以及對比學習基礎模型MedImageInsight。通過在Azure AI平臺上部署這些先進的多模態醫療AI模型,微軟旨在為醫療行業提供全新的工具,推動醫療服務的智能化發展。
作者簡介
論文的五位共同一作及通訊作者均為華人,分別來自微軟和華盛頓大學。
趙正德(TheodoreZhao),論文第一作者,為該研究作出主要技術貢獻。微軟高級應用科學家,現主要研究方向包括多模態醫療AI模型,圖像分割與處理,大模型的安全性分析(Pareto最優誤差估計)。本科畢業于復旦大學物理系,博士畢業于華盛頓大學應用數學系,期間研究希爾伯特-黃變換和分數布朗運動的多尺度特征,以及隨機優化在醫療領域的應用。

顧禹(AidenGu),論文共同一作,微軟高級應用科學家,致力于推動AI在醫療領域的發展。本科畢業于北京大學微電子與經濟專業。其研究方向專注于醫療健康、生物醫學,以及機器人多模態模型。代表性工作包括創建首個領域特定的大語言模型PubMedBERT,以及患者旅程模擬模型BiomedJourney。

潘海峰(HoifungPoon),論文通訊作者,微軟研究院健康未來(Health Futures)General Manager,華盛頓大學(西雅圖)計算機博士。研究方向為生成式AI基礎研究以及精準醫療應用。在多個AI頂會獲最佳論文獎(比如NAACL,EMNLP,UAI),在HuggingFace上發布的開源生物醫學大模型總下載量達數千萬次(比如PubMedBERT,BioGPT,BiomedCLIP,LLaVA-Med),在《自然》上發表首個全切片數字病理學模型 GigaPath(自五月底公布以來已被下載四十萬次),部分研究成果開始在合作的醫療機構和制藥公司中轉化為應用。

王晟(ShengWang),論文通訊作者,華盛頓大學計算機科學與工程系助理教授,專注于人工智能與醫學的交叉研究,利用生成式AI解決生物醫學問題。他的科研成果已在《Nature》《Science》《Nature Biotechnology》《Nature Methods》和《The Lancet Oncology》等頂級期刊上發表十余篇論文,并被Mayo Clinic、Chan Zuckerberg Biohub、UW Medicine、Providence等多家知名醫療機構廣泛應用。

Mu Wei,論文通訊作者,微軟Health and Life Sciences團隊首席應用科學家,擁有十余年醫療與金融領域的AI模型研發與部署經驗。他的團隊聚焦于健康領域的多模態AI模型,研究成果涵蓋生物醫學圖像解析、數字病理學基礎模型、臨床文檔結構化的大模型應用以及大模型錯誤率估計等方向。

論文地址:https://www.nature.com/articles/s41592-024-02499-w
項目展示網頁:https://microsoft.github.io/BiomedParse/
GitHub:https://aka.ms/biomedparse-release
數據集:https://huggingface.co/datasets/microsoft/BiomedParseData
AzureAPI網頁:Model catalog - Azure AI Studio
- 免責聲明
- 本文所包含的觀點僅代表作者個人看法,不代表新火種的觀點。在新火種上獲取的所有信息均不應被視為投資建議。新火種對本文可能提及或鏈接的任何項目不表示認可。 交易和投資涉及高風險,讀者在采取與本文內容相關的任何行動之前,請務必進行充分的盡職調查。最終的決策應該基于您自己的獨立判斷。新火種不對因依賴本文觀點而產生的任何金錢損失負任何責任。





 新火種
2024-11-21
新火種
2024-11-21